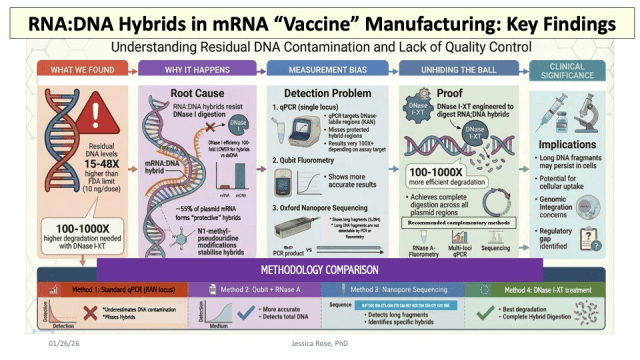
L’infographie signée par Jessica Rose, PhD (janvier 2026), résume des conclusions d’une recherche récente (préprint ou preprint publié fin 2025) sur la contamination par ADN résiduel dans les vaccins à ARNm COVID-19 (Pfizer et Moderna).
Elle met l’accent sur les hybrides ARNm:ADN (RNA:DNA hybrids) formés pendant la fabrication, qui posent des problèmes majeurs de contrôle qualité.Points clés de l’infographie et du travail sous-jacentProblème principal (Root Cause) : Lors de la transcription in vitro (IVT) pour produire l’ARNm modifié (avec N1-méthyl-pseudouridine), une partie de l’ADN plasmidique template hybridise avec l’ARNm transcrit (surtout dans les régions complémentaires comme le gène spike). Ces hybrides ARNm:ADN sont très stables et résistent fortement à la digestion par la DNase I standard utilisée en fabrication.
Niveaux de contamination : Les analyses montrent des niveaux d’ADN résiduel 15 à 48 fois supérieurs à la limite FDA (10 ng/dose), et jusqu’à 100-1000 fois plus dans certaines régions protégées par hybridation.
Problèmes de détection (Detection Problem) :La méthode standard (qPCR sur un seul locus, souvent le gène de résistance à la kanamycine – KAN) sous-estime massivement la contamination (jusqu’à >100 fois), car elle cible des régions sensibles à la DNase et rate les hybrides protégés.
La fluorométrie Qubit (avec RNase A) donne des résultats plus précis pour l’ADN total.
Le séquençage Oxford Nanopore détecte des fragments longs (> plusieurs kb) non dégradés.
Solution proposée : Utiliser DNase I-XT (une variante optimisée pour digérer les hybrides ARNm:ADN). Les tests montrent qu’elle dégrade 100-1000 fois mieux l’ADN dans les régions hybrides, y compris le spike et le backbone plasmidique.
Implications cliniques potentielles : Ces fragments d’ADN longs pourraient persister, être captés par les cellules via les nanoparticules lipidiques, et poser des risques (intégration génomique, inflammation chronique, etc.). Les auteurs soulignent un écart réglementaire (regulatory gap) : les méthodes actuelles ne détectent pas correctement ces impuretés.
Ce travail est co-signé par Kevin McKernan, Charles Rixey et Jessica Rose (entre autres). Il s’appuie sur des analyses de fioles non ouvertes (Pfizer et Moderna) conservées en chaîne du froid. Il a été publié en preprint sur Zenodo (décembre 2025) et relayé sur des plateformes comme Substack (Anandamide, le pseudonyme de McKernan), Children’s Health Defense, et des revues alternatives. Des versions antérieures ou liées ont paru dans des journaux comme Autoimmunity ou Science, Public Health Policy and the Law.
